GraphSAGE
Inductive Representation Learning on Large Graphs
NIPS 2017 GraphSAGE
主要贡献:
- 提出了一种inductive学习图结构的方法,使用节点的特征(文本、度等)作为输入,进行无监督学习
- 构造了三个节点分类(node classification)的数据集
1 Introduction
从transductive到inductive 在GCN中训练需要训练整个图,所有节点的embedding,之后基于这些训练过的embedding进行测试,这叫做transductive; GraphSAGE计划设计一个inductive的方法,不训练所有的节点。在测试集中的节点在训练时是不可见的,使用节点的特征作为原始输入,然后使用训练好的网络在测试集中评估。
提出了GraphSAGE(SAmple and aggreGAte)
2 Method

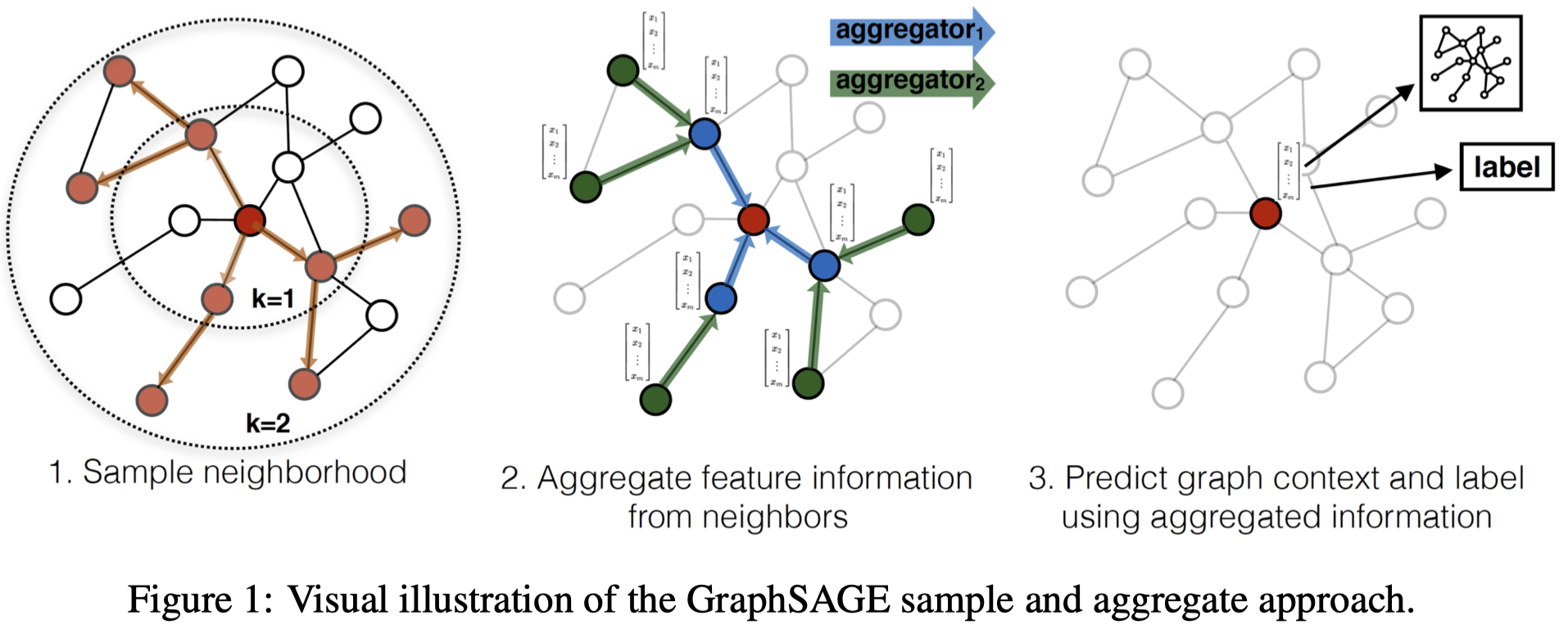
核心在于如何聚合邻居节点的信息:
- 首先使用节点特征(文本等)作为初始输入
- 之后均匀随机采样固定数量的直接相邻邻居节点,定义特定的聚合函数AGGREGATE,聚合邻居信息(不包括自身的特征)
- 拼接自身的特征与邻居的信息,过一层神经网络,得到获得一阶邻居后的输出
- 上面的过程重复K次,每个节点在k次聚和时,采样\(S_k\)个邻居的k-1阶表示,得到k阶表示
损失函数: \[ J(\mathbf{z}_u) = -log(\sigma(\mathbf{z}_u^T \mathbf{z}_v))-Q\mathbb{E}_{v_n\sim P_n(v)}log(\sigma(-\mathbf{z}_u^T \mathbf{z}_{v_n})) \] \(v\)是邻居节点;\(v_n\)是负样本;\(P_n(v)\)是负采样分布;\(Q\)是负采样的数量。这个loss的含义是让邻居节点相似,增大不相关的节点差异。
下面是GraphSAGE的核心方法,聚合函数。
在论文里提出了四个聚合函数:
- -GCN:\(\mathbf{h}_v^k = \sigma(W\cdot \mbox{Mean}(\{\mathbf{h}_v^{k-1}\} \cup \{\mathbf{h}_u^{k-1},\ u\in \forall N(v) \} ))\)
- -Mean:先平均邻居表示,之后与中心节点表示拼接后过一层神经网络,\(\mbox{AGGREGATE}^{\mbox{mean}}_k=\mbox{Mean}(\{\mathbf{h}_u^{k-1},\ u\in \forall N(v) \})\)
- -LSTM:使用LSTM聚合邻居信息,每次聚合时先随机打乱邻居节点的顺序
- -Pooling:\(\mbox{AGGREGATE}^{\mbox{pool}}_k=\mbox{max}(\{\sigma(W_{pool}\mathbf{h}_u^{k-1}+\mathbf{b}),\ u\in \forall N(v) \})\)
3 Experiments
三个实验:
- Web of Science引文网络分类任务,判断paper属于哪个subject。
- Reddit中发送的post的分类任务,判断用户发送的post属于哪个community
- protein-protein interaction (PPI)分类任务,判断蛋白质的功能
对应的构造了三个数据集
- Wos Data:从Web of Science Core Collection中收集,2000-2005年6个生物领域的论文,标签就是这6个生物领域。使用2000-2004年数据作为训练,使用30%的2005年数据作为验证集,70%作为测试集。最终数据集包括了302,424节点,平均degree 9.15。在实验时,使用论文abstract的sentence embedding以及节点的degree作为初始特征输入。
- Reddit Data:从Reddit 2014年9月中构造数据,选择了50个大的Reddit communities,如果用户在两个post下进行了评论,就把这两个post连接起来。20天训练,剩下的3天验证,7天测试。最终包含232,965节点,平均度492。实验时,post的title embedding,comment embedding,post的打分,comment的数量作为初始特征输入。
- PPT Data:从Molecular Signatures Database中构造数据集,20个graph训练,2个graph验证,2个graph测试。每个graph来自不同的人体组织,平均graph有2373个节点,平均degree 28.8,一共121个标签。
在验证集上寻找各个模型最好的参数,然后在测试集上评估。
实验时,还对比了监督学习(直接与标签进行cross-entropy)和无监督学习

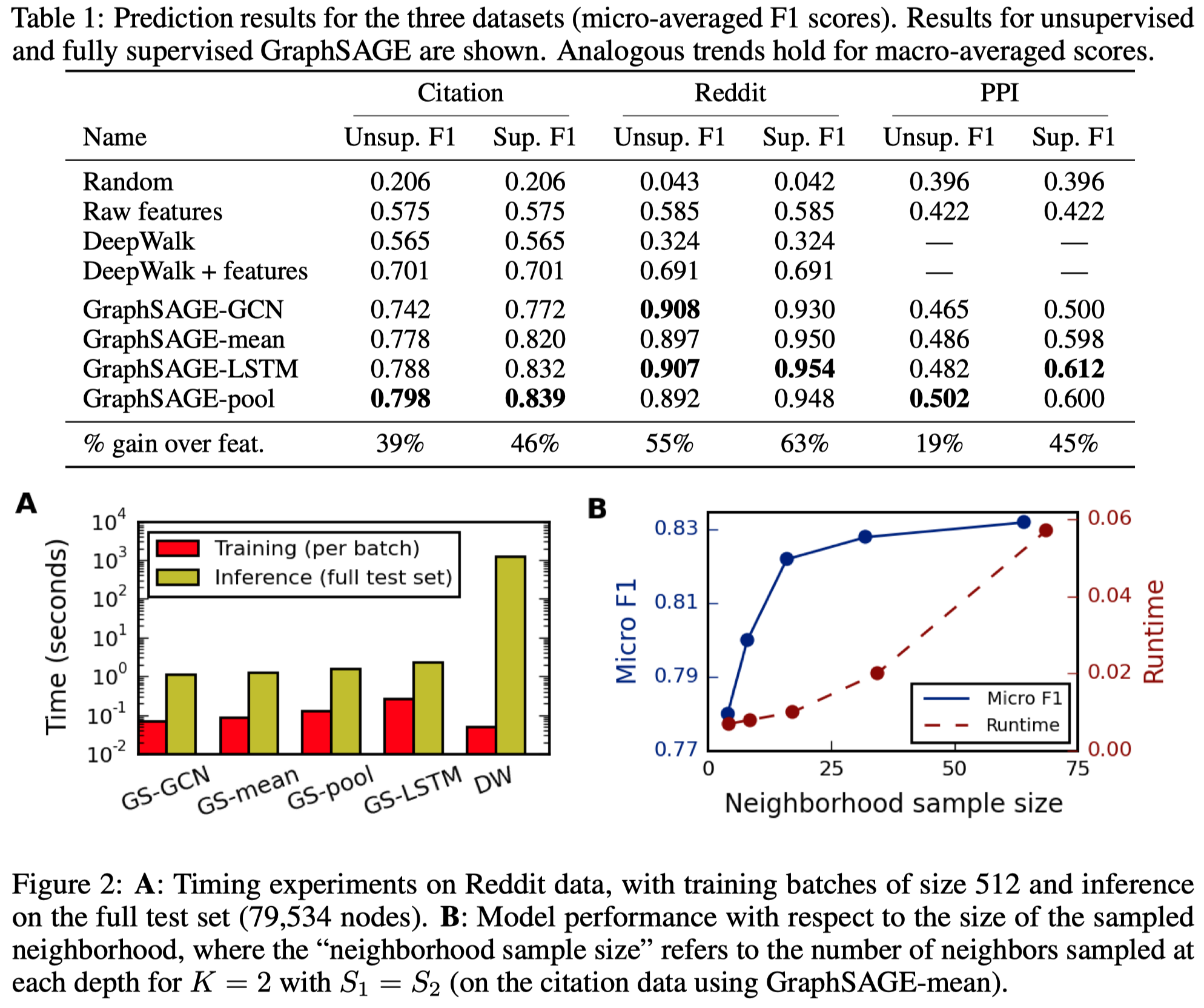
最终,作者发现K=2相对是比较好的选择,同时,采样s邻居数量\(S_1\cdot S_2 < 500\)较好,实验时使用的GraphSAGE都是K=2,\(S_1=25\),\(S_2=10\)。
4 Minibatch pseudocode
首先采样在对batch \(B\)进行K阶训练,需要用到的所有节点。\(B^k\)包括了所有在训练\(k+1\)时需要用到的节点的\(k\)阶表示。